Flavoproteine
Flavoproteine stellen einen Großteil der Proteine aus Organismen.
Flavine
Flavine sind die am häufigsten anzutreffenden Kofaktoren von Proteinen in der Natur. Sie können drei Redox-Zustände annehmen: voll oxidiert, ein-Elektron-reduziert und voll reduziert. Jeder dieser Redox-Zustände existiert in einer kationischen, neutralen und anionischen Form, abhängig vom pH-Wert der Umgebung. Der voll oxidierte Redox-Zustand ist typischerweise der energetisch stabilste in Flavoproteinen.
Flavine absorbieren Licht im sichtbaren Wellenlängenbereich: Die optische Absorption des Grundzustands der Flavine liegt zwischen 400 und 500 nm. Zusammen mit der hohen Quantenausbeute für das Intersystem Crossing (ISC) und das hohe Oxidationspotential des Triplett-Zustandes ist diese Eigenschaft für die Photooxidation der Flavine verantwortlich.
Diese Eigenschaften sind ebenfalls Voraussetzung für die quantitative Generierung des Semichinon-Radikals und der Hydrochinon-Form der Flavine in Proteinen ausgehend vom voll oxidierten Zustand. Diese Photoreduktions-Reaktionen laufen typischerweise unter Blaulicht ab. Sie konnten auch ohne externe Elektronendonatoren in Proteinen, z.B. der DNA-Photolyase, beobachtet werden.
Blaulichtaktive Flavoproteine
Blaulichtaktive Flavoproteine lassen sich in drei Klassen einteilen:
Phototropine
Die Phototropine und homologen Proteine (Phot-Proteine) sind am Phototropismus (dem gerichteten Pflanzenwachstum in Richtung einer Lichtquelle), der Chloroplastenbewegung, der Stomata-Öffnung in Schließzellen der Blätter, der schnellen Inhibitoion des Sproßwachstums und der Gametogenese (sexuelle Differenzierung in Algen) beteiligt.
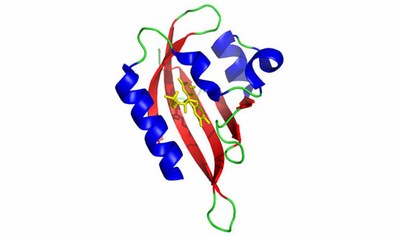
Abbildung: Kristallstruktur der Phot-LOV1-Domäne von Chlamydomonas reinhardtii im Dunkelzustand. Daten aus: Fedorov, R., Schlichting, I., Hartmann, E., Domratcheva, T., Fuhrmann, M., Hegemann, P.: Crystal Structures and Molecular Mechanism of a Light-Induced Signaling Switch: The Phot-LOV1 Domain from Chlamydomonas reinhardtii, Biophys. J. 84:2474, 2003. PDB ID: 1N9L. Abbildung erstellt mit PyMOL
Literatur
- E. Schleicher, R.M. Kowalczyk, C.W.M. Kay, P. Hegemann, A. Bacher, M. Fischer, R. Bittl, G. Richter, S. Weber
On the reaction mechanism of adduct formation in LOV domains of the plant blue-light receptor phototropin
Journal of the American Chemical Society 126 (2004) 11067-11076 - C.W.M. Kay, E. Schleicher, A. Kuppig, H. Hofner, W. Rüdiger, M. Schleicher, M. Fischer, A. Bacher, S. Weber, G. Richter
Blue light perception in plants. Detection and characterization of a light-induced neutral flavin radical in a C450A mutant of phototropin
Journal of Biological Chemistry 278 (2003) 10973-10982
BLUF-Domänen enthaltende Proteine
Rezeptoren mit BLUF-Domänen agieren als Redox- und Licht-regulierte Derepressoren in Purpurbakterien oder als Photorezeptoren für die Steuerung von Verhaltensreaktionen in Flagellaten wie Euglena.
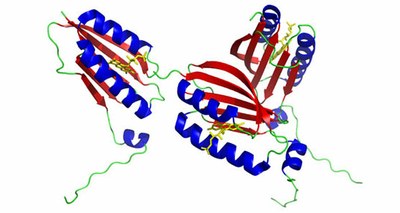
Abbildung: Kristallstruktur der BLUF-Domäne von AppA aus Rhodobacter sphaeroides. Daten aus: Anderson, S., Dragnea, V., Masuda, S., Ybe, J., Moffat, K., Bauer, C.: Structure of a Novel Photoreceptor, the BLUF Domain of AppA from Rhodobacter sphaeroides. Biochemistry 44:7998, 2005. PDB ID: 1YRX. Abbildung erstellt mit PyMOL
Photolyasen / Cryptochrome
Die Photolyase/Cryptochrom Proteinfamilie besteht aus einer Reihe untereinander stark homologer Proteine:
- Photolyasen, die UV-Schäden an der DNA reparieren und ihrerseits nach zu reparierendem Photoprodukt in CPD- und 6,4-Photolyasen unterteilt werden,
- Cryptochrome als Blaulicht-Rezeptoren, die sowohl im Pflanzen- als auch im Tierreich verbreitet und an der Regulation des circadianen Rhythmus beteiligt sind, und
- DASH-Cryptochrome, strukturell den Photolyasen am ähnlichsten, aber ohne DNA-Reparaturaktivität, deren Funktion bisher unbekannt ist.
Für die EPR-Spektroskopie interessant ist die Tatsache, daß alle diese Proteine nach Lichtanregung des Flavin-Kofaktors im blauen Wellenlängenbereich ein Radikalpaar-Signal in der transienten EPR zeigen, das dem Flavin einerseits und einer Aminosäure (Trp oder Tyr) andererseits zugeordnet werden kann.
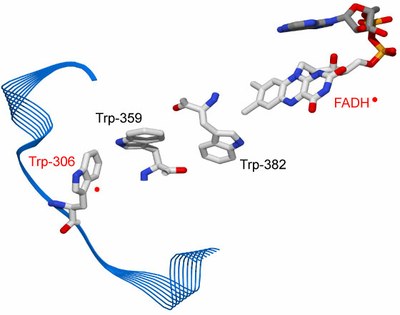
Abbildung: Tryptophan-Kette in Escherichia coli CPD-Photolyase, die am Elektronentransport vom Trp-306 zum Flavin (FAD) beteiligt ist. Rot markiert sind hier die beiden Partner des Radikalpaars, das an der Proteinoberfläche gelegene terminale Tryptophan (Trp-306) und der im Protein verborgene Flavin-Kofaktor (FAD).
Literatur
- S. Weber
Light-driven enzymatic catalysis of DNA repair: a review of recent biophysical studies on photolyase
Biochimica et Biophysica Acta (Bioenergetics) 1707 (2005) 1-23 - C.W.M. Kay, H. Mögling, E. Schleicher, K. Hitomi, K. Möbius, T. Todo, A. Bacher, G. Richter, S. Weber
A comparative time-resolved electron paramagnetic resonance study of the flavin cofactor photoreduction in Escherichia coli cyclobutane pyrimidine dimer photolyase and Xenopus laevis (6-4) photolyase
Flavins and Flavoproteins 2002 (S. Chapman, R. Perham, N. Scrutton, Editors), Rudolf Weber Agency for Scientific Publishing, Berlin (2002) 713-718 - S. Weber, C.W.M. Kay, H. Mögling, K. Möbius, K. Hitomi, T. Todo
Photoactivation of the flavin cofactor in Xenopus laevis (6-4) photolyase: observation of a transient tyrosyl radical by time-resolved electron paramagnetic resonance
Proceedings of the National Academy of Sciences USA 99 (2002) 1319-1323 - S. Weber, G. Richter, E. Schleicher, A. Bacher, K. Möbius, C.W.M. Kay
Substrate binding to DNA photolyase studied by electron paramagnetic resonance
Biophysical Journal 81 (2001) 1195–1204 - S. Weber, K. Möbius, G. Richter, C.W.M. Kay
The electronic structure of the flavin cofactor in DNA photolyase
Journal of the American Chemical Society 123 (2001) 3790-3798
Proteine aller drei Klassen werden mit unterschiedlichen Methoden der EPR untersucht.

